BGZToolkit
BGZIP 是生物信息学领域的常用压缩工具,它将输入文件切分成近似 64KB 的小块,并单独应用 Deflater 压缩成一系列小的 BGZF 块,这使得可以针对压缩后的块构建索引信息,并用于快速地检索部分数据,而无需解压整个文件。
本项目使用纯 Java 脚本开发 BGZ 文件的操作方法,包括并行压缩、解压、拼接、文件校验等功能。
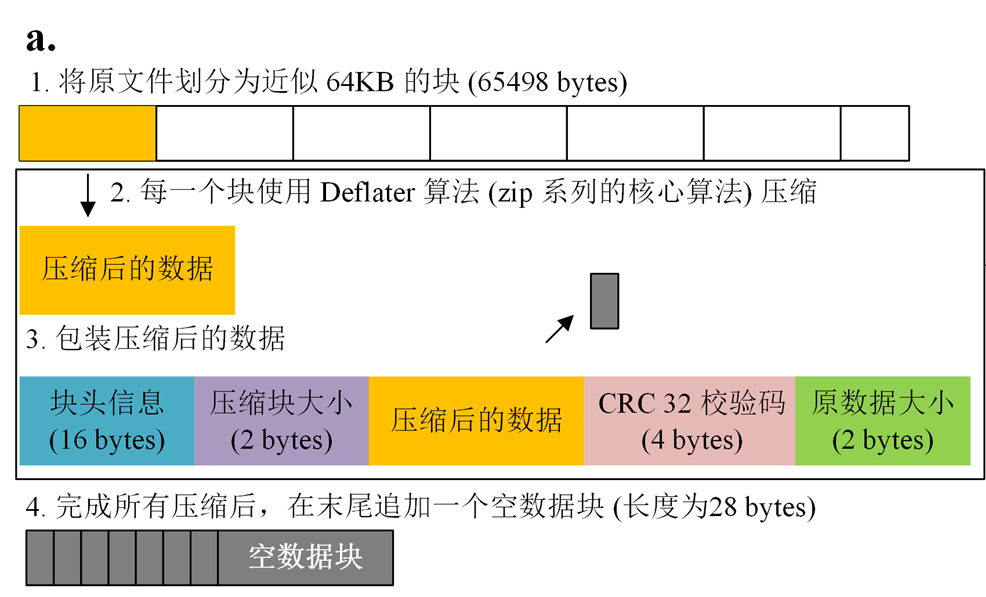
API 方法
BGZIP 的主要 API 方法可以在 edu.sysu.pmglab.bgztools.BGZToolkit 中找到 (现已集成在 commandParser 包中),分别为:
- 压缩文件: BGZToolkit.Compress.instance(File inputFile, File outputFile)
- 设置并行压缩线程数: .setThreads(int nThreads)
- 设置压缩的文件范围: .limit(long start, long end)
- 设置压缩级别: .setCompressionLevel(int compressionLevel)
- 提取文件: BGZToolkit.Extract.instance(File inputFile, File outputFile)
- 设置并行压缩线程数: .setThreads(int nThreads)
- 设置读取的文件范围: .limit(long start, long end)
- 设置输出文件格式: .setOutputParam(BGZOutputParam outputParam)
- 计算文件的 MD5 码: BGZToolkit.MD5.instance(File inputFile)
- 是否计算解压后文件的 MD5 码: .setDecompression(boolean decompression)
- 连接多个子文件: BGZToolkit.Concat.instance(File[] inputFiles, File outputFile)
实例化任务完成后, 通过 .submit() 提交相应的任务.
根据 API 设计解析器
BGZIP 工具集共有 5 个独立运行模式: 压缩、解压、连接、MD5 检查、MD5 检查 (解压信息),总共有包括线程数、文件指针范围、输出文件、压缩级别、是否打印日志在内的 5 个参数。创建步骤如下:
Step1: 创建参数组:Mode 组, Options 组;
Step2: 创建 Mode 组参数: --compress; --decompress; --concat; --md5; --md5-decompress;
Step3: 创建 Options 组参数: --output; --range; --level; --threads;
Step4: 设定参数规则:
- Mode 组参数 --compress; --decompress; --concat; --md5; --md5-decompress 必须传入一个;
- 选定了 --md5 或 --md5-decompress 模式时, 无法使用 --output; --range; --level; --threads 参数;
- 选定了 --concat 模式时, 无法使用 --range; --level; --threads 参数;
Step5: 设定程序名为: <mode>;
Step6: 导出 Java Script Builder With Options Format 格式文件。
设计主函数
使用 CommandParser 桥接用户参数与业务逻辑:
public static void main(String[] args) {
try {
BGZToolkitParser options = BGZToolkitParser.parse(args);
if (options.getOptions().isHelp()) {
System.out.println(BGZToolkitParser.usage());
return;
}
if (options.compress.isPassedIn) {
// 压缩文件
long startTime = System.currentTimeMillis();
File outputFile = options.output.isPassedIn ? options.output.value : options.compress.value.addExtension(".gz");
Compress task = Compress.instance(options.compress.value, outputFile)
.setThreads(options.threads.value)
.setCompressionLevel(options.level.value)
.setPrintLog(true);
if (options.range.isPassedIn) {
task.limit(options.range.value[0], options.range.value[1]);
}
task.submit();
logger.info("Compression completed. Total time: {} s; File size: {}", (System.currentTimeMillis() - startTime) / 1000d, outputFile.formatSize(3));
return;
}
if (options.decompress.isPassedIn) {
long startTime = System.currentTimeMillis();
// 解压文件
File outputFile;
Extract task;
if (options.output.isPassedIn) {
outputFile = options.output.value;
task = Extract.instance(options.decompress.value, outputFile)
.setThreads(options.threads.value);
if (options.level.isPassedIn || options.output.value.withExtension(".gz")) {
task.setOutputParam(new BGZOutputParam(options.level.value));
}
} else {
if (!options.level.isPassedIn) {
// 没有传入 level 时, 认为是解压文件
outputFile = options.decompress.value.changeExtension("", ".gz");
if (outputFile.equals(options.decompress.value)) {
logger.error("can't remove an extension from {} -- please rename", options.decompress.value);
return;
}
} else {
// 传入了 level, 认为是提取文件子集
if (options.decompress.value.withExtension(".gz")) {
logger.error("missing required positional argument: --output");
return;
} else {
outputFile = options.decompress.value.addExtension(".gz");
}
}
task = Extract.instance(options.decompress.value, outputFile)
.setThreads(options.threads.value);
}
if (options.range.isPassedIn) {
task.limit(options.range.value[0], options.range.value[1]);
}
task.submit();
logger.info("Decompression completed. Total time: {} s; File size: {}", (System.currentTimeMillis() - startTime) / 1000d, outputFile.formatSize(3));
return;
}
if (options.concat.isPassedIn) {
// 连接文件
long startTime = System.currentTimeMillis();
if (!options.output.isPassedIn) {
logger.error("missing required positional argument: --output");
return;
}
Concat.instance(options.concat.value, options.output.value).submit();
logger.info("Concat completed. Total time: {} s; File size: {}", (System.currentTimeMillis() - startTime) / 1000d, options.output.value.formatSize(3));
return;
}
// md5 校验文件
if (options.md5.isPassedIn) {
MD5 task = MD5.instance(options.md5.value);
task.setDecompression(false);
System.out.println("MD5 (" + options.md5.value + ") = " + task.submit());
return;
}
if (options.md5Decompress.isPassedIn) {
MD5 task = MD5.instance(options.md5Decompress.value);
task.setDecompression(true);
System.out.println("MD5 (" + options.md5Decompress.value + ") = " + task.submit());
return;
}
} catch (Exception | Error e) {
logger.error("{}", e.getMessage(), e);
}
}
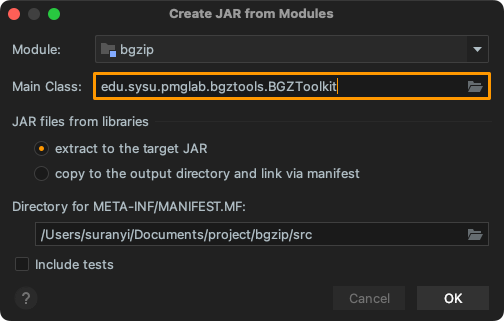
创建 jar 包
依次点击: Project Structure… > Artifacts > + > JAR > From modules with dependencies …, 在 Main Class 处选择入口函数位置,并打包为:
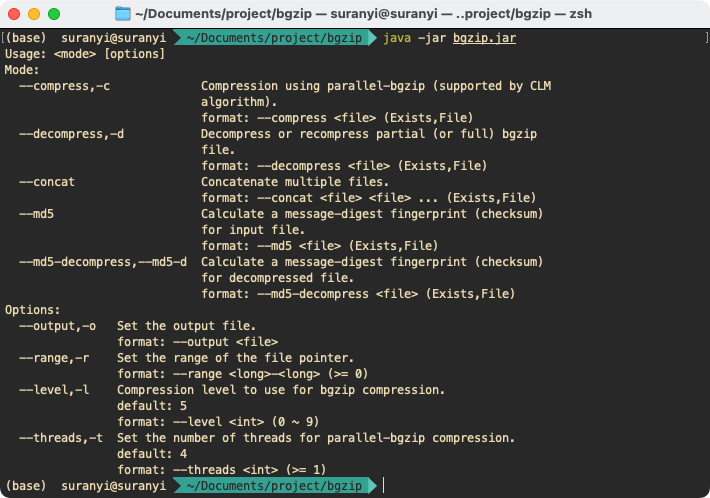
进入 bgzip.jar 所在路径,在控制台输入指令显示文档:
java -jar bgzip.jar
应用 BGZToolkit
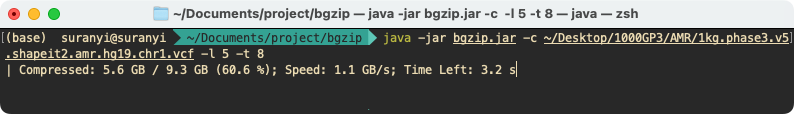
1. 多线程压缩单个文件
# 文件来源: http://pmglab.top/genotypes/#/
java -jar bgzip.jar -c ~/Desktop/1000GP3/AMR/1kg.phase3.v5.shapeit2.amr.hg19.chr1.vcf -l 5 -t 8
2. 解压单个文件,并重新压缩为 bgzip 格式
# 提取前 1 GB 内容
java -jar bgzip.jar -d ~/Desktop/1000GP3/AMR/1kg.phase3.v5.shapeit2.amr.hg19.chr1.vcf.gz \
--range 0-1073741824 -o ~/Desktop/subFile.vcf.gz
# 解压上述文件
java -jar bgzip.jar -d ~/Desktop/subFile.vcf.gz
3. 检验文件 MD5 码
# 结果: 29ccebe9d2748328f6cc8b16c6563261
java -jar bgzip.jar --md5 ~/Desktop/subFile.vcf
# 结果: 29ccebe9d2748328f6cc8b16c6563261
java -jar bgzip.jar --md5-d ~/Desktop/subFile.vcf.gz
4. 连接多个文件
# 先拆分出 2 个文件
java -jar bgzip.jar -d ~/Desktop/1000GP3/AMR/1kg.phase3.v5.shapeit2.amr.hg19.chr1.vcf.gz \
--range 0-536870912 -o ~/Desktop/subFile1.vcf.gz
java -jar bgzip.jar -d ~/Desktop/1000GP3/AMR/1kg.phase3.v5.shapeit2.amr.hg19.chr1.vcf.gz \
--range 536870912-1073741824 -o ~/Desktop/subFile2.vcf.gz
# 拼接两个子文件
java -jar bgzip.jar --concat ~/Desktop/subFile1.vcf.gz ~/Desktop/subFile2.vcf.gz -o ~/Desktop/subFile.merge.vcf.gz
# 校验 md5 码, 结果: 29ccebe9d2748328f6cc8b16c6563261
java -jar bgzip.jar --md5-d ~/Desktop/subFile.merge.vcf.gz