合并多个 GTB
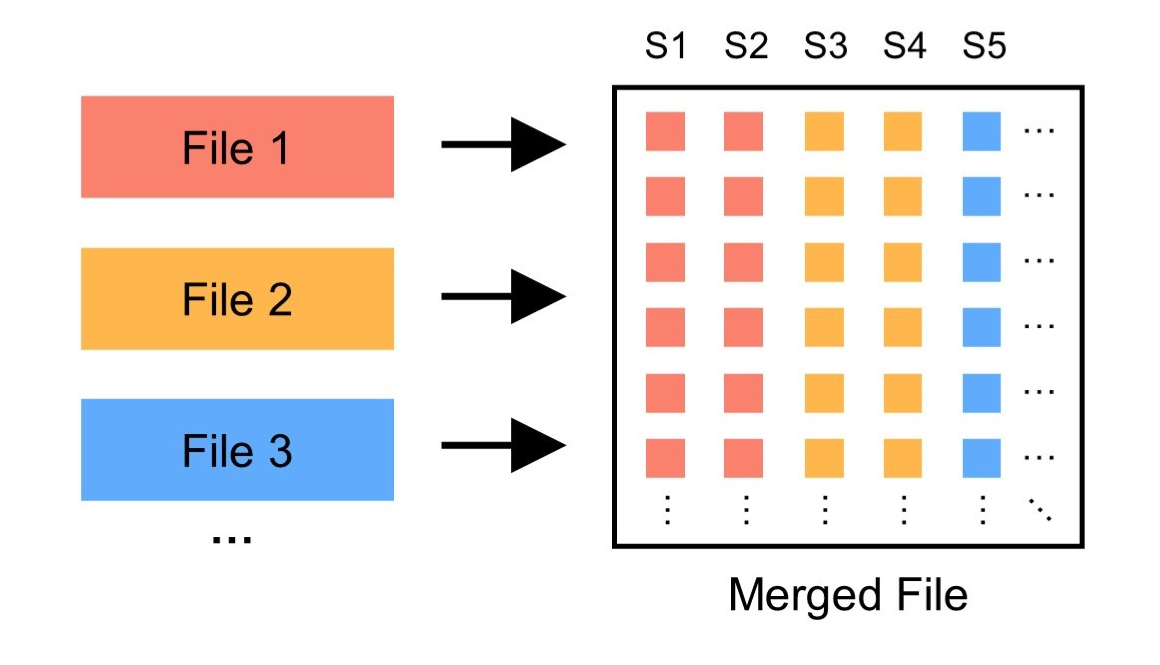
合并即建立覆盖多个输入文件的样本超集(例如:将 1000GP3 中的不同群体的基因组文件合并为单独的文件),也包括将相同坐标的字段相互组合。由于多个文件的合并在非基因型上相当复杂(例如,某些统计字段的更新、注释信息的合并),因此命令行工具中 GBC 仅提供基因型的合并,使用以下指令对 GTB 文件进行基因型合并:
java -jar gbc.jar merge <input> <input> ... -o <output> [options]
合并后的新文件可以通过 GTBExporter 指令进行拆分。GBC 的文件合并采用两两合并的策略(使用以样本量为权重的最小堆进行优化),以适应任意规模样本集间的合并。
[!NOTE|label:示例程序|style:callout]
使用 GBC 合并 1000GP3 的所有 Y 染色体基因型:
# 下载文件 wget https://pmglab.top/gbc/download/1000GP3.hg19.chrY/afr.gtb \ https://pmglab.top/gbc/download/1000GP3.hg19.chrY/amr.gtb \ https://pmglab.top/gbc/download/1000GP3.hg19.chrY/eas.gtb \ https://pmglab.top/gbc/download/1000GP3.hg19.chrY/eur.gtb \ https://pmglab.top/gbc/download/1000GP3.hg19.chrY/sas.gtb # 在终端直接运行 java -jar gbc.jar merge ./afr.gtb ./amr.gtb ./eas.gtb ./eur.gtb ./sas.gtb \ -o ./1000GP3.chrY.gtb # 使用 docker 运行 docker run -v `pwd`:`pwd` -w `pwd` --rm -it -m 4g gbc \ merge ./afr.gtb ./amr.gtb ./eas.gtb ./eur.gtb ./sas.gtb \ -o ./1000GP3.chrY.gtb
程序参数
语法: merge <input> <input> ... -o <output> [options]
Java-API: edu.sysu.pmglab.gbc.toolkit.GTBMerger
关于: 合并多个 GTB 文件中的受试者基因型.
参数:
*--output,-o 设置输出文件.
格式: --output <file>
--chromosome 指定染色体标签文件.
格式: --chromosome <file>
--threads,-t 设置并行线程数.
默认值: 4
格式: --threads <int>
--method,-m 处理多个文件坐标的方式 (合并、交集、与第一个文件对齐).
默认值: alignment
格式: --method <string> ([union/intersection/alignment] (忽略大小写))
--no-gt 不加载和储存基因型, 仅合并坐标和等位基因.
--add-meta 添加元信息到输出文件.
格式: --add-meta <key>=<value> <key>=<value> ...
--rm-meta 移除所有元信息.
API 工具
对 GTB 文件进行合并的 API 工具是 edu.sysu.pmglab.gbc.GTBMerger,使用示例如下:
GTBMerger.of("./afr.gtb", "./amr.gtb")
.setMergeOperator(GTBMerger.MergeOperator.UNION)
.submit();
在 API 工具中,通过 addField 添加额外字段名和字段类型、 addValueConverter 设置新字段值,实现非基因型字段的合并。