连接多个 GTB
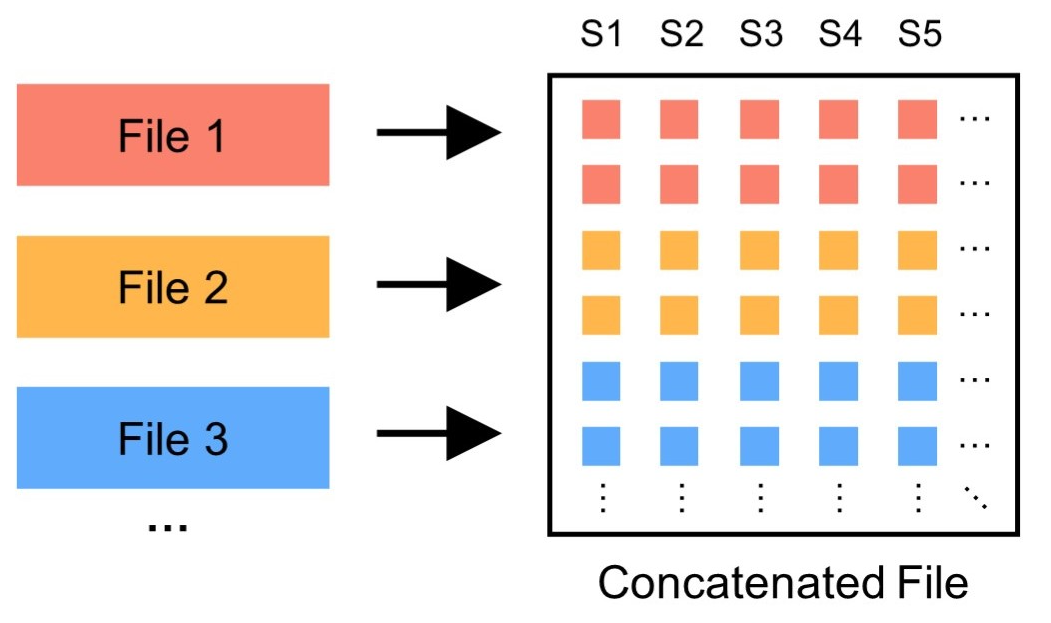
连接即按行添加新的变异位点,通常这些位点之间的坐标不重叠。最常见的情形是将分散在不同文件(按照染色体、按照变异位点数量、文件大小储存)的同一批受试者的变异位点重新拼接回单独的文件(例如:将 1000GP3 中的 chr1~chr22, chrX, chrY 拼接为单独的文件)。使用以下指令对 GTB 文件进行连接:
java -jar gbc.jar concat <input> <input> ... -o <output> [options]
多个文件进行拼接时,GBC 会检查每个子文件的第一个位点并进行排序,以确保拼接后的文件尽可能保持 “坐标有序”。
[!NOTE|label:示例程序|style:callout]
使用 GBC 将 GTBSplitter 产生的多个子文件进行连接:
# 在终端直接运行 java -jar gbc.jar concat ./rare.disease.hg19/chr5.gtb ./rare.disease.hg19/chr7.gtb ./rare.disease.hg19/chr8.gtb ./rare.disease.hg19/chr10.gtb ./rare.disease.hg19/chr17.gtb ./rare.disease.hg19/chr21.gtb -o ./rare.disease.hg19.concat.gtb # 使用 docker 运行 docker run -v `pwd`:`pwd` -w `pwd` --rm -it -m 4g gbc \ concat ./rare.disease.hg19/chr5.gtb ./rare.disease.hg19/chr7.gtb ./rare.disease.hg19/chr8.gtb ./rare.disease.hg19/chr10.gtb ./rare.disease.hg19/chr17.gtb ./rare.disease.hg19/chr21.gtb -o ./rare.disease.hg19.concat.gtb
[!TIP|label:拼接一个文件夹下所有的 GTB 文件|style:callout]
在 Macos、Linux 的终端环境下运行时,可以使用以下语句列出 “DATA_PATH” 文件夹下的所有以
.gtb为扩展名的文件并以空格连接。$(find $DATA_PATH -type f -name '*.gtb' -exec echo {} \;)例如,上述示例程序与以下程序相同:
# 在终端直接运行 java -jar gbc.jar concat $(find ./rare.disease.hg19 -type f -name '*.gtb' -exec echo {} \;) -o ./rare.disease.hg19.concat.gtb
程序参数
语法: concat <input> <input> ... -o <output> [options]
Java-API: edu.sysu.pmglab.gbc.toolkit.GTBConcat
关于: 连接多个 GTB 文件.
参数:
*--output,-o 设置输出文件.
格式: --output <file>
--chromosome 指定染色体标签文件.
格式: --chromosome <file>
--threads,-t 设置并行线程数.
默认值: 4
格式: --threads <int>
--field,-f 筛选连接到输出文件的字段. 所选择的字段必须在所有子文件中都存在.
格式: --field <string>,<string>,...
--subject,-s 指定连接到输出文件的受试者 (基因型). 受试者名称可以以逗号分隔的格式存储在文件中,并通过'-s @file'传入. 默认连接所有受试者的基因型. 当受试者在某个子文件中不存在时, 它的基因型被设置为 './.' (或 '.|.', 取决于向型).
格式: --subject <string>,<string>,...
--no-gt 不加载和储存基因型, 仅连接坐标和补充字段.
--sort 按照坐标 (CHROM, POS) 排序变异位点.
--add-meta 添加元信息到输出文件.
格式: --add-meta <key>=<value> <key>=<value> ...
--rm-meta 移除所有元信息.
API 工具
对 GTB 文件进行连接的 API 工具是 edu.sysu.pmglab.gbc.GTBConcat,使用示例如下:
GTBConcat.of(new File("./rare.disease.hg19"))
.addManager("./rare.disease.hg19/chr5.gtb")
.addManager("./rare.disease.hg19/chr7.gtb")
.addManager("./rare.disease.hg19/chr8.gtb")
.addManager("./rare.disease.hg19/chr10.gtb")
.addManager("./rare.disease.hg19/chr17.gtb")
.addManager("./rare.disease.hg19/chr19.gtb")
.addManager("./rare.disease.hg19/chr21.gtb")
.submit();